Bulk-sequencing 혹은 Spatial transcriptomics 데이터의 Phenotype을 보기 위해,
Histopathology (혹은 Whole-slide Imaging) 데이터와 결합하여 보는 경우가 많아졌다.
나는 이 연구를 시작하게 된 계기는, 하버드 Peter park 랩에서 Normal tissue에 대한 CNV 연구가 활발한데, 정상인에서 발견되는 초기암으로 생각되는 CNV의 phenotype을 WSI에서 과연 볼 수 있을까? 에 대한 주제로 코웍을 하게 된 것인데,
만약 Histopathology에서 CNV를 어느정도 탐지할 수 있는 능력이 있다면, 때 초기암의 phenotype으로 여겨지기도 하는 hyperplasia같은 영역을 중요한 patch로 꼽지 않을까? 하는 질문에 관한 연구이다.
나는 주로 Multiple Instance Learning (MIL) 을 통해 이 문제를 해결하고자 했다.
이 글을 쓰는 이유는, histopathology를 다루기 위한 MIL이라는 메소드의 가능성, 그리고 한계에 대한 것을 정리하기 위함이고, 앞으로 공부하고자 하는 GNN을 어느정도 정리하고 싶어서이다.
1. Multiple Instance Learning (MIL)
MIL은 하나의 Label이 부여된 데이터에, 다중의 데이터가 포함되어있는 것을 처리하기 위한 메소드이다.
Instance: 각 데이터를 의미
Bag: 여러개의 Instance를 가지는 모음
이 때, Bag에 정답에 해당하는 label이 부여된다.
다양한 경우가 있겠지만, Bag 마다 존재할 수 있는 Instance가 상이할 수 있기에, 정형데이터만을 다루는 메소드로는 이 문제에 당야한 문제를 불러올 수 있습니다. 따라서, 여러개으 Instance로부터 추출된 정보를 'Aggregation' 하는 방법이 매우 중요하게 여겨지는 문제라고 볼 수 있습니다.
Histopathology에서의 MIL이라고 한다면, Bag은 하나의 WSI를 나타내며, Instance는 그 안의 Patch라고 할 수 있습니다.
WSI가 너무 크기때문에, 이를 한번에 처리할 수 있는 CNN을 구현하는 것은 사실상 불가능하며, 따라서 Patch에서 feature를 추출하여 이를 통합하는 메소드가 현재 MIL의 정석적인 방법과도 같이 여겨집니다.
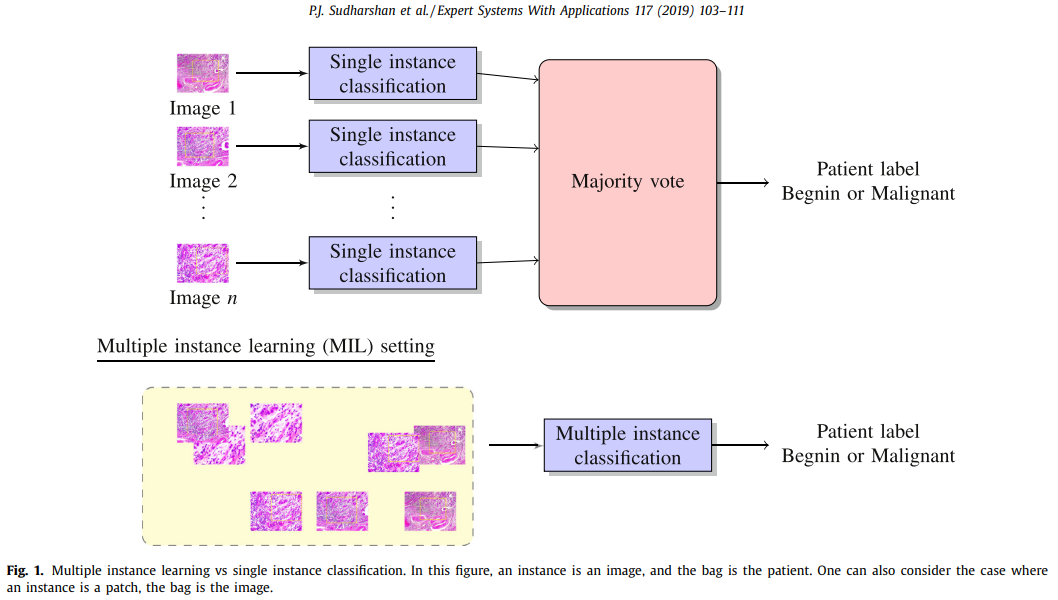
최근 주요했던 Histopathology를 다루는 메소드중 하나인 하버드 faisal lab에서 나온 CLAM을 설명하면 다음과 같다.
- Clustering constrained Attention MIL (CLAM)
Lu, Ming Y., et al. "Data-efficient and weakly supervised computational pathology on whole-slide images." Nature biomedical engineering 5.6 (2021): 555-570.
CLAM은 Label을 예측하는데 도움이 되는 'discrimitive regions of interest'를 clustering-based attention mechanism을 통해서 예측하는 메소드. 비슷한 feature를 가지는 patch들을 그룹으로 묶어, cluster를 기반으로 weight를 부여하여 최종 bag-level label을 예측하는 것을 목표로 합니다.
아래 디테일한 clustering-based attention mechanism을 제외하면, MIL을 통해 histopathology를 다루는 논문들은 거의 비슷한 workflow를 가진다.


CLAM 연구에서는 256*256*3 image를 input으로 받아, ResNet50 모델의 3번째 Residual block의 output에 대하여 mean-spatial pooling을 적용하여, 1024개의 feature vector를 얻었다.
여기서 MIL의 단점이 드러나는데, Feature를 extracting 하는 과정에서 사용되는 weight는 end-to-end style로 fine-tuning 되지 않기에, imageNet pretrained model이 'Pathology image를 잘 분해할 것이라는 가정' 이 필요하다. 이를 해결하기 위해 feature의 분해능을 training 과정에서 좋게하기 위한 semi-supervised 등의 메소드 (왜냐하면 각 instance들은 weak-supervision이며, distinct한 feature를 가지는 instance들의 차별화가 bag-label의 학습에 도움이 될 것이라는 가정) 들이 응용된 논문들이 몇 보이는데
모든 MIL 메소드가 비슷하듯, 각 instance의 weight를 가늠할 수 있는 메소드를 활용하여, 최종적으로 시각화 한다.

Attention score를 통해 heatmap을 그리는 것은 단순하지만, Attention은 어떻게 구하는지? 에 관해서는 이 문제를 근본적으로 다뤘던 페이퍼인 아래의 페이퍼를 따라가면 된다. (2018년도에 이런 논문을.. 정말 대단한 것 같다)
Ilse, Maximilian, Jakub Tomczak, and Max Welling. "Attention-based deep multiple instance learning." International conference on machine learning. PMLR, 2018. (이 페이퍼에서 제공하는 코드는 정말 이해하기 쉽게 구현되어있다.)

내가 그려본 "Attention-based deep multiple instance learning." 논문의 Worflow scheme이다.
위 그림에서 'Feature Extraction' 과 'Attention mechanism' 두가지 개념이 가장 중요하다.

- Feature extractor
CNN을 활용하여, 중간 Feature를 뽑는 것이다. 다만, CNN layer의 output은 Unit (N) * Matrix (2차원) 에 해당하는 3차원 데이터이기 때문에, 각 Matrix를 'Global Average Pooling' 등을 활용하여 차원을 축소하여 점 (0차원) 으로 만들어야 한다.
즉, 최종 Vector (1d) 처럼 만들어야 하기에, N개의 Matrix로 이루어진 CNN의 output을, pooling 하여야 한다.
- Attention mechanism
가장 기본적인 Linear regression의 경우 (Y = aX + b), Input X에 대하여 coefficient 'a' 가 Y에 미치는 영향으로 해석할 수 있다.
이를 확장하여, 다차원의 Input data가 들어오는 경우를 생각해보자.
다차원의 데이터를 처리하기 위한 Layer는 linear regression 처럼 바로 해석가능한 coefficient (weight, parameter) 를 가질 수 없기 때문에, 다차원의 Input Xk에 대하여 0차원의 point value로 바꾸는 network를 생성한다.
해당 network의 output과 Input Xk에 곱연산을 통해, 데이터 자체에 Attention을 부여하는 식의 네트워크를 생각할 수 있다.
상단에서 제시한 위 Workflow scheme에서, ('Workflow scheme of Attention based Deep Multiple Instance Learning') Extracted feature vectors (각각이 Xk) 에 Attention weight가 곱해져서 그 다음 Layer에 넘어가는 것을 확인할 수 있다.
학습이 잘 되면 될 수록, (흰색으로 옅어지는) Output에 영향을 주지 못하는 Input Xk는 weight가 작아져서 filter-out 될 것이며, (검은색으로 굵게 되는) Output에 영향을 잘 주는 Xk는 weight가 높아져서 filter-in 될 것이다. 이를 통해서 Multiple Input에 대한 중요도를 구할 수 있다. 이것이 바로 Attention 개념이다.
Summary, Multiple Instance Learning
MIL 기술에 대해 정리를 해보면, 현재까지 사용되고 있는 Histopathology 데이터를 다루기 위한 MIL은 다음과 같다.
다차원의 Input의 feature로 추출하여, Matrix (Row: Patch, Column: Extracted Features) 형태의 데이터를 활용하여 Pathology level에서의 Label을 맞추는 것.
뭔가 어려워보이지만, 단순 Matrix 데이터를 활용하여 Label에 맞추는 것에 불과해보이기도 한다. 여기서의 쟁점은, Attention mechanism을 활용하여 어떤 Patch가 (row가) 중요했냐를 추론하여, Histopathology 상에서 중요한 부위를 검증하고, 이를 pathology knowledge와 연결하는 것이다.
Limitation of MIL
한계는 명확하다. Feature extactor가 pathology image의 texture-feature를 매우 잘 구분할 수 있을 것이라는 가정이 필요하다. Feature extraction Feature selection과 modeling이 나뉘어져있던 과거의 machine learning의 시대처럼, 이는 최적의 model을 학습하기가 어려울 것이라는 것을 암시한다는 걸 우리는 알 수 있다.
MIL 논문들을 읽어보면, 대체로 Cancer type classification, Cancer vs Normal의 분류, Metastasis가 존재하는 Tissue 찾기 등, Pathology에 전문지식이 없는 일반 사람들도 몇 분 남짓한 짧은 교육으로 충분히 수행할 수 있는 task 들이다. 즉 매우 매우 쉬운 일들만 objective로 활용하여 기술들이 나오고 있다는 것을 의미한다.
Breast Tissue에서 MIL을 활용하여 Gender를 예측하는 것 정도는 초등학생도 알려주면 금방 할 수 있는 일이다. 나의 경험 상 이런 정도의 task에서는 MIL이 매우 매우 잘 동작한다. 하지만 Esophagus 처럼 성별에 따른 차별적인 feature가 잘 보이지 않는 Pathology image에서는 MIL을 활용해서는 구분이 매우 매우 어렵다. 전문가라면 Epithelial tissue의 범위, 그리고 connective tissue 등의 spatial한 정보를 통해 대충 짐작 가능하다만, MIL을 통해 이를 예측하는 것은 현재의 기술로는 사실상 쉽지가 않다. 왜냐하면, Esophagus sample에서 export pathologist가 성별을 구분하는 원리를 MIL에서 사용되는 Feature extractor가 전혀 데이터를 이해하지 못하고 단순 Texture feature를 뽑아내기 때문이다.
Tissue의 광범위한 texture feature를 활용하여 유전자의 변이, CNV 등을 막연히 딥러닝으로 때려박아서 AUC가 0.7정도 달성했다는 논문들은 많지만, 아직은 시기상조인것 같다. AUC가 0.7이라도 나오는 이유는, 개인적인 생각으로는 KRAS같은 유전적 변이를 texture feature로부터 찾은 것이 아니라, 암의 Stage, Severity가 KRAS 변이와 같은 유전적 이벤트와 상당한 Association (혹은 correlation)이 있기 때문인 것 같다.
2. Graph Neural Network (GNN)
Graph structure 데이터는 'Entity' 들의 복잡한 관계나 Interaction을 설명하는데 매우 유용하다. Graph 자체는 Entitiy를 Node로써, 그리고 관계 (relationships) 를 'Edge' 로 정의하여 활용할 수 있다.
Pathology image를 다루기 위한 GNN에서, 가장 기본적인 Terminology를 정의해보자.
Nodes: Biological structures within tissue (Cells)
Edges: Relationships between nodes (Spatial proximity)
Graph: A set of nodes and graphs
이러한 Graph 구조를 Neural network에 넣기 위해서는, Matrix 구조로 바꾸어야 한다.
- Adjacency matrix: Node간의 relationship에 관한 데이터

Adjacency matrix는 각 Node간의 Relationship에 관한 데이터를 담을 수 있다.
(Row: node, Column: relationship between nodes)
각 Node는 또한 Feature를 가질 수 있다.
(Row: node, Column: Features)
기본적인 GNN은 2가지를 Input으로 받는 것을 예로 들 수 있다.
[Adjacency matrix, Feature matrix]
* Propagation Rule
자, 위의 E에 해당하는 Adjacency matrix에는 4개의 Node가 서로 Relationship을 나타내는 데이터가 있다.
이에, 2개의 feature를 가지는 Feature matrix가 있다고 가정하자.
Propagation은 이 두가지, Adjacency matrix와 Feature matrix의 곱으로 표현할 수 있다.
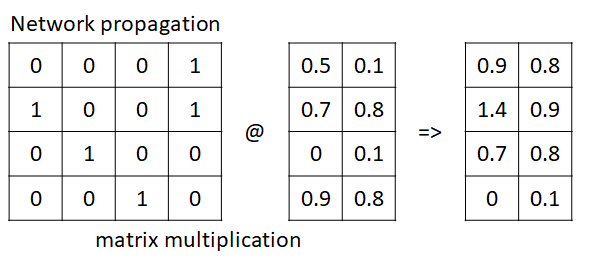
위의 Matrix multiplication을 잘 생각해서 보자.
첫번째 노드는, 4가지의 노드에 대하여 0, 0, 0, 1의 관계를 가지고 있는데, 각 4가지 노드는 중간 matrix의 feature들을 갖고 있다.
이들간의 matrix multiplication은,
[(0*0.5, 0*0.7, 0*0.0, 1*0.9),
(0*0.1, 0*0.8, 0*0.1, 1*0.8)] 로 나타낼 수 있는데,
사실 이걸 잘 생각해보면 첫번째 Node와 연결되어있는 모든 Node의 feature의 합성곱으로 생각할 수 있다.
물론 Network의 weight에 따라, 연결되어있는 노드 feature에 가중치가 곱해져서 더해지는 것으로 볼 수 있다.
따라서, 우리는 각 Node feature에 대해서 연결되어있는 Node들간의 feature에 대해서 정보를 propagation (전파) 하는 것으로 생각할 수 있다.
이를 공식으로 나타내면 다음과 같다.
f(Hⁱ, A) = σ(AHⁱWⁱ)
H: Feature map
A: Adjacency matrix
W: Weight matrix
물론 위의 예는 단순 propagation이며, 이들 사이에 Weight가 부여되면, 이것이 바로 Graph neural network가 되는 것이다.
Propagation 하는 과정에서 skip-connection을 추가하여 ResNet과 비슷한 특성을 띄게 할 수 있으며, 또한 Attention machenism도 이 과정에서 부여할 수 있다.
이처럼, node간의 관계인 Adjacency matrix와 Node feature를 나타내는 Feature matrix 2가지를 input으로 하여, 우리가 원하는 Whole-slide level에서 학습을 시도할 수 있다.
다음 그림은 AI를 활용하여 Pathology image를 분석하는 유명한 스타트업인 PathAI의 Webinar 발표자료에서 인용하였다.

Histopathology에서 Cell, Tissue에 관한 데이터를 추출한다.
하지만 Node가 너무 많아지면, 하드웨어 한계상 연산이 불가능하기에, Clustering을 통하여 Spatial information을 적절하게 갖도록 한다.
그 이후에, GNN을 통하여 최종적으로 알고자 하는 Label을 예측하도록 학습할 수 있다.
##
Whole slide image 라는 엄청난 사이즈의 이미지를 다루기 위한 두가지 기술을 공부해봤다.
Multiple instance learning과 Graph Neural Network,
두가지 모두 여전히 연구수준에서 사용되는 기술이며, 모두 장단점을 가지고 있다.
앞으로 많은 연구들에서 활용할 수 있을 듯 하다.
'Major Study. > Bioinformatics' 카테고리의 다른 글
| DINO Contrastive Learning in Medical Imaging (2) | 2023.11.17 |
|---|---|
| Cross-validation 평균 ROC 구하기 (R/Python) (1) | 2023.08.25 |
| Single Cell Analysis Best Practice 정리해보기 (1) | 2023.02.06 |
| GTEx에서 Pathology image 분석하기 (0) | 2022.10.13 |
| LUAD의 Lymph meta를 Radiomics, Deep Learning으로 비교 (0) | 2022.10.08 |