Normal H&E Slide image를 분석할 일이 생겨서,
분석을 해본 겸, 정리 포스트를 남겨놓으려고 한다.
1. GTEx phenotype 데이터 활용하기
개인적으로 TCGA, ICGC, GTEx 등 유전체 데이터를 활용할 때, UCSC Xena를 자주 활용한다.
서로 다른 데이터베이스의 batch effect 등을 정리한 데이터까지 제공해서.. 무척 편하다.
phenotype - GTEX phenotype (n=9,783) UCSC Toil RNA-seq Recompute
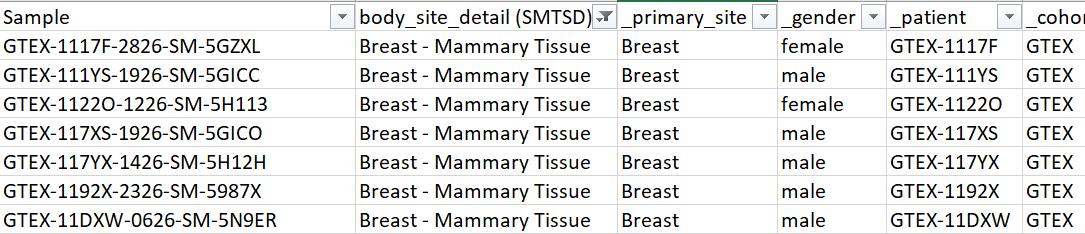
GTEx 데이터에서 원하는 tissue의 sample ID를 가져올 수 있다.
나는 이번 pathology image 분석에서는 breast tissue를 활용하고자 했는데,
다음과 같이 phenotype data에서 sample들을 확인할 수 있다.
(Whole Slide Image의 용량이 좀 커서 전부다 다운받지는 않았다)
2. GTEx portal에서 histology viewer 에서 샘플 확인하기
https://gtexportal.org/home/histologyPage
GTEx Portal
gtexportal.org
아래와 같이 원하는 sample을 클릭 하면, pathology image를 확인할 수 있다.
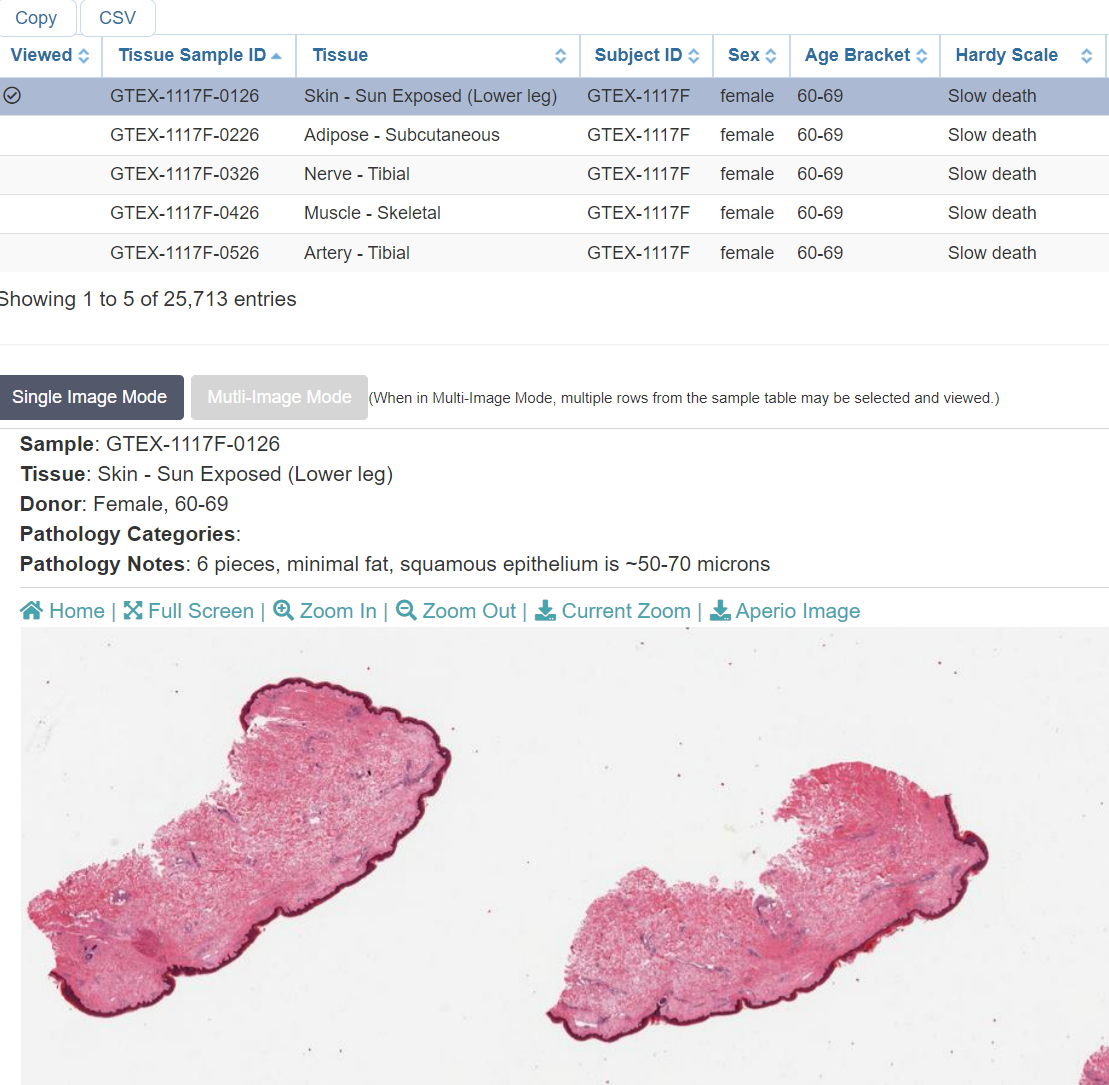
위 slide image에서 Aperio Image 버튼이 보이는데, URL을 확인해보면 다음과 같다.
https://brd.nci.nih.gov/brd/imagedownload/GTEX-1117F-0126
가장 뒤에 GTEx 샘플의 이름을 넣게 되면, 해당 slide image를 다운받을 수 있다.
1번에서 받은 phenotype 데이터에서, sample list를 위 주소와 조합하여
wget으로 쭈르륵 받으면 끝!
3. Patch (tile) 뜯어내기
openslide가 대표적인 pathology image 분석 tool이지만,
단순히 tile만 뜯어내면 되는 작업이었어서, py-wsi 라는 패키지를 활용하였다.

Magnification을 최고화질로 하여, patch를 모두 저장하게되면 용량이 만만치 않으므로,
py-wsi 패키지의 저장관련 함수를 직접 수정하여, 중요한 patch만 저장한다.
(Brightness, 혹은 Red-color의 비중 등을 활용하여 의미있는 patch 인지를 체크)

밝기로만 patch를 컨트롤 하게되면, 그림자가 있거나 quality가 안좋은 tissue도 다수 포함될 수 있으니 주의..
'Major Study. > Bioinformatics' 카테고리의 다른 글
| Histopathology를 다루기 위한 MIL (1) | 2023.02.26 |
|---|---|
| Single Cell Analysis Best Practice 정리해보기 (1) | 2023.02.06 |
| LUAD의 Lymph meta를 Radiomics, Deep Learning으로 비교 (0) | 2022.10.08 |
| Multi-class와 Multi-label classificiation, 분포가 다른 Output들 (0) | 2022.08.18 |
| deep learning for the life sciences (3) | 2022.06.12 |