CCLE database review
Cancer Cell Line Encyclopedia (CCLE)
- to conduct a detailed genetic and pharmacologic characterization of a large panel of human cancer models to develop integrated computational analyses that link distinct pharmacologic vulnerabilities to genomic patterns and to translate cell line integrative genomics into cancer patient stratification.
The CCLE provides public access to genomic data, analysis and visualization for about 1000 cell lines.
현재 기준으로 1046개의 cell line data가 있음.
1. Data
http://www.broadinstitute.org/ccle/data/browseData?conversationPropagation=begin
1) DNA Copy Number (41.6GB) Affy SNP (Published)
- Affymetrix SNP6.0 arrays.
Compressed archive of the raw CEL files
Raw Affymetrix CEL files were converted to a single value for each probe set representing a SNP allele or a copy number probe.
- Copy number
Copy numbers were then inferred based upon estimating probe set specific linear calibration curves, followed by normalization by the most similar HapMap normal samples.
Segmentation of normalized log2 ratios (specifically, log2(CN/2)) was performed using the circular binary segmentation (CBS) algorithm.
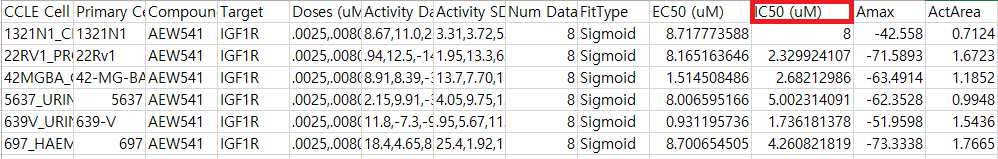
2. Pharmacological profiling (8.0MB) Drug data (Published)
Pharmacologic profiles for 24 anticancer drugs across 504 cell lines.
Cell line, Drug, target gene, 그리고 dose에 따른 activity, 그리고 IC50 값이 주어진다.
3. mRNA expression (8.0GB) Gene expression (Published)
Affymetrix U133+2 arrays.
Raw Affymetrix CEL files were converted to a single value for each probe set using Robust Multi-array Average (RMA) and normalized using quantile normalization.
raw CEL 파일도 제공하지만, 이미 RMA로 normalization이 다 되어있기 떄문에 다루기는 매우 쉬울 것 같다.
Entrez 이름이 있는 gene-centric expression data를 받아서 보면 좋을 것 같다.
- SNP and Expression data from the Cancer Cell Line Encyclopedia (CCLE)
http://www.ebi.ac.uk/arrayexpress/files/E-GEOD-36139/
ebi array
4.1 Oncomap mutations (464.8KB) Mutation (Published)
The mutations were assessed in 33 genes (381 specific mutations) using Oncomap 3.0 core
4.2 Hybrid capture sequencing (6.5GB) Mutation (In process)
List of mutations and indels in 1651 genes, determined by targeted massively parallel sequencing
maf, csv로 매우 쉽게 제공된다.
대충 보니, dbSNP_RS가 cell line을 나타내는 column인 듯 싶다.
데이터 다루기에 매우 쉽고 간결하게 제공해주고 있다.
4.3 binary calls for copy number and mutation data (95.9MB) Mutation (In process)
Copy number 데이터도 제공된다.
GDSC database처럼, binary 형태로 제공된다.
데이터는 Nature에 publush 되었고, 지금까지 1343회 인용되었음.
위 논문에서 CCLE 데이터베이스를 이용해서 데이터를 분석 한 후,
mutation & copy number & expression 상태에 따라서 24개의 anticancer-drug를 prioritization 해주는 논문
Figure1에서는 우습게도 Panobinostat이 모든 tissue type에서 제일 좋았다고 한다 ㅋㅋ.
아마 제일 독한 약이 Genomic features와 관계 없이 IC50 값이 가장 낮았기 때문에 그럴 것이라 생각.
SVM, WEKA 등을 이용해서 데이터들을 분석한다.
Sensitive: IC50 <= 0.5uM
Restant: IC50 > 0.5uM
으로 두 군을 나눈것은 눈여겨 볼 만 할것 같다.
Taxane, Platinum 약물을 쓰는 ovarian cancer study에서, sensitive vs resistant 두 그룹으로 나눈 GEO 데이터가 존재한다.
이 논문에서는 sensitive, resistant 두 그룹의 DEG를 구하고 sensitive에서 과발현되는 유전자, resistant에서 과발현되는 유전자 목록을 구한다.
TCGA 데이터와 GEO 데이터에서 위에서 찾은 DEG 유전자 발현량을 통해 drug resistance에 대한 scoring 메소드를 적용.
- ssGSEA (single sample Gene Set Enrichment Analysis)
이것이 실제로 생존을 유의하게 나누었다고 통계수치를 이용하여 자신들의 논리를 주장.
Drug response associated gene expression을 가지는 gene들은 Cell cycle, plasma membrane에 enriched 되어있다고 한다. 또한 targeting sex specific cancer drug가 알려져있다고 함.
이 논문에선 young / old sample간의 Drug response associated gene expression을 밝힘..
아이디어는 정말 단순하다.
gene expression과 drug sensitivity의 통계분석, Ontology analysis, sex specific Drug response pattern, age specific drug response pattern 등을 연구하여 정리한 논문
안녕하세요 포스팅 잘 읽었습니다.
혼자서 공부중인 학생인데요ㅠ
혹시 지금 CCLE데이터에서는 IC50값을 다운받으려면 어떤 파일을 다운받아야 하는지 알수있을까요?
https://depmap.org/portal/download/
여기 파일 대부분을 다운받아봤는데도 없어서 이렇게 댓글 남깁니다
아무리 찾아도 없어서요ㅠㅠ 도움주시면 정말정말 감사드리겠습니다ㅠㅠㅠ
## 6년 전에 썼던 글에 반가운 댓글이 달려서 오랜만에 저도 접속해봤습니다.
BroadInstitute의 CCLE 데이터가 Depmap에 통합되었네요,
=> Select a dataset to view: 를 클릭하신 후,
'Drug Screens' 중에 Pharmacological profiling를 누릅니다.
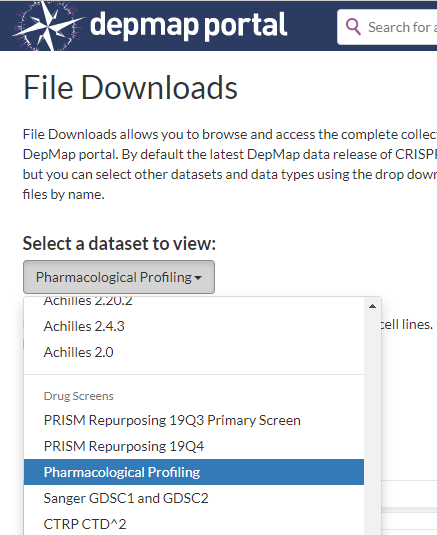
그리고 Drug_data 다운받으시면 됩니다. 벌써 CCLE가 7년이 넘었네요..